Investigadores del Centro Nacional de Biotecnología (CNB) del CSIC han publicado en la revista Nature Communications un estudio que muestra cómo la sensibilidad colateral puede inducirse de forma temporal, algo no descrito hasta la fecha, para evitar la aparición de cepas bacterianas resistentes a los antibióticos y para mejorar la respuesta terapéutica de fármacos antibacterianos ya existentes.
Se trata de la sensibilidad colateral transitoria’, llamada así por los investigadores del CNB-CSIC firmantes del estudio Sara Hernando-Amado, Pablo Laborda y José Luis Martínez. Si la sensibilidad colateral es la compensación evolutiva por la que la adquisición de una mutación de resistencia a un antibiótico por parte de una bacteria conlleva mayor sensibilidad a otro, el tipo de sensibilidad colateral descrita por los investigadores destaca por ser inducible de forma temporal, por evitar la aparición de mutaciones de resistencia a los antibióticos en las bacterias tratadas y por proporcionar una mejora terapéutica en el uso de antibióticos ya comercializados.
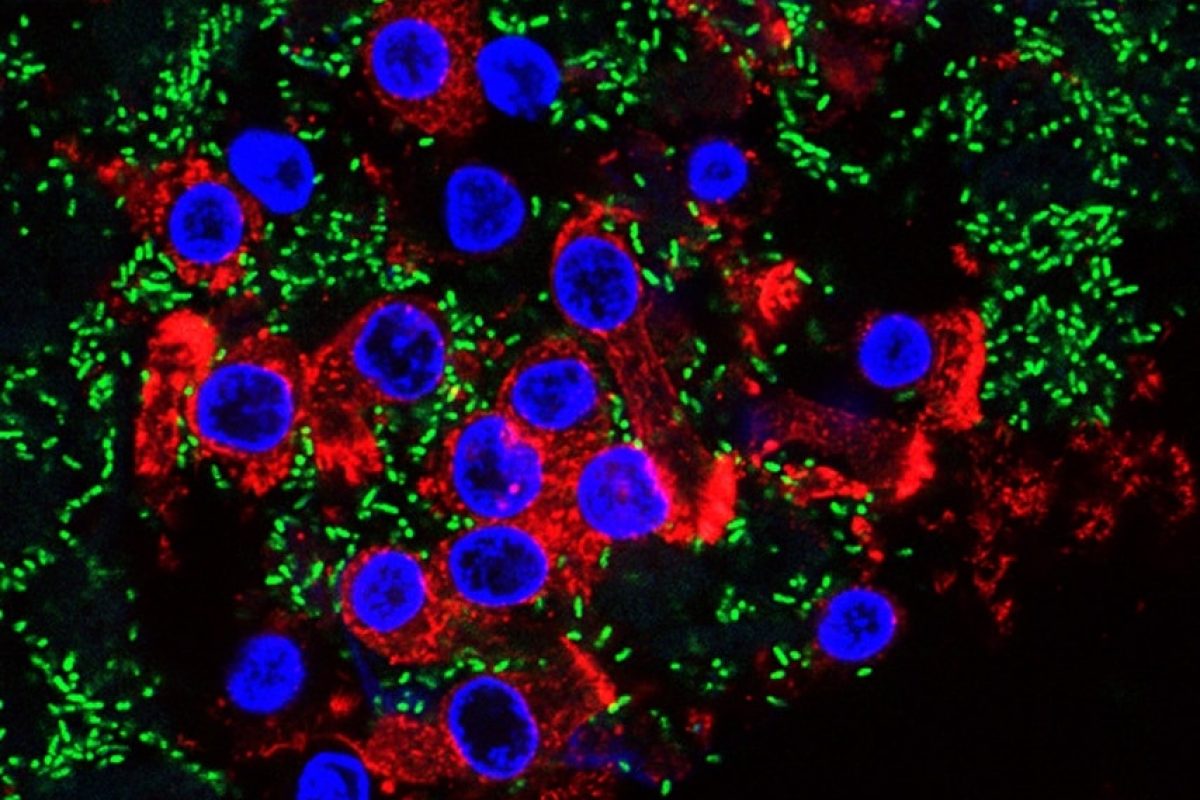
El equipo del CNB-CSIC liderado por José Luis Martínez ha demostrado que se puede inducir la sensibilidad colateral utilizando cloruro de decualinio, un fármaco con propiedades antisépticas y desinfectantes. Este compuesto, a diferencia de los antibióticos, no selecciona mutaciones de resistencia en diferentes cepas mutantes y cepas clínicas multirresistentes, pero induce sensibilidad colateral transitoria, de forma consistente, en dichas cepas. En estudios previos, los autores identificaron la aparición de sensibilidad colateral al antibiótico tobramicina tras el uso de ciprofloxacino, y que esta sensibilidad era mayor cuando se adquirían mutaciones en un gen concreto (nfxB). Por otro lado, el equipo identificó que este compuesto era capaz de inactivar NfxB de forma temporal, produciendo una resistencia transitoria (inducible) al antibiótico ciprofloxacino.
A ello se suman los resultados del presente estudio, en el que los investigadores muestran que esta resistencia transitoria también lleva asociada la sensibilidad colateral a tobramicina. La investigadora que ha liderado el trabajo en el CNB-CSIC, Sara Hernando-Amado, explica que “si bien el fenómeno de la sensibilidad colateral ha sido profundamente estudiado, nunca se había planteado que fuera posible inducirla temporalmente, de forma no heredable, y de modo robusto en diferentes mutantes y cepas clínicas resistentes a diferentes antibióticos”.
Además, continúa la investigadora, “esta estrategia supone una mejora en el tratamiento que propusimos recientemente, donde explotábamos la sensibilidad colateral clásica, dado que inducimos un mayor nivel de sensibilidad a tobramicina. Además, la resistencia que provoca el cloruro de decualinio a ciprofloxacino es transitoria, desaparece cuando lo hace este compuesto, de modo que en el proceso no se acumulan nuevas mutaciones estables de resistencia bacteriana, como ocurre con los tratamientos clásicos”.
Sensibilidad sin resistencia
“Nos preguntamos si inhibiendo de forma temporal la actividad de NfxB (induciendo así una resistencia transitoria) podríamos también dar lugar a un estado de sensibilidad transitoria a tobramicina, algo que podríamos explotar terapéuticamente y que evitaría la selección de mutantes resistentes a un primer antibiótico”, explica Hernando-Amado.
Los resultados del grupo de José Luis Martínez muestran que el uso combinado de cloruro de decualinio y tobramicina elimina tanto cepas mutantes resistentes de la bacteria Pseudomonas aeruginosa (un patógeno prevalente que causa infecciones en pacientes hospitalizados o con patologías previas y que genera infecciones crónicas en pacientes con fibrosis quística) como cepas clínicas muy variadas, incluyendo las inicialmente resistentes a tobramicina, y sin causar la adquisición de mutaciones genéticas que favorezcan la resistencia a otros antibióticos.
Este trabajo demuestra que el uso de este compuesto y de otros aún por identificar podría permitir el diseño de nuevas estrategias terapéuticas para tratar las infecciones bacterianas.
Fagos para inducir la muerte de bacterias
Por otro lado, también vinculado a las infecciones provocadas por bacterias resistentes a antibióticos, un grupo de investigación del Instituto de Biología Integrativa de Sistemas (i2SysBio), centro mixto del Consejo Superior de Investigaciones Científicas (CSIC) y la Universitat de València (UV), ha desarrollado una molécula basada en los bacteriófagos o fagos, virus que matan bacterias, para provocar la muerte de estas por despolarización del citoplasma, que hace que las células de las bacterias no mantengan la carga eléctrica para llevar a cabo sus funciones vitales y mueran irreversiblemente.
Una de las terapias alternativas más prometedoras a los antibióticos convencionales son los bacteriófagos o fagos. Son virus que infectan y parasitan bacterias, y suponen las entidades biológicas más abundantes del planeta. Cada fago es específico de un determinado género o especie bacteriana, lo que permite dirigirse contra una bacteria específica. Actúan como otros virus: se unen a un receptor existente en la superficie bacteriana e inyectan su material genético en su interior, se replican y la destruyen.
Sin embargo, “las bacterias tienen un sistema de defensa que también las puede hacer resistentes a los fagos”, argumenta Alfonso Jaramillo, investigador del CSIC en el I2SysBio. Su laboratorio de Biología Sintética De Novo acaba de comenzar un proyecto para desarrollar una molécula imitando unas que ya existen en la naturaleza y que se parece a un fago, pero que no lo es. Aunque se conocían estas moléculas nunca había sido posible evolucionarlas, lo que es necesario para matar a bacterias de interés. “Se trata de fagos sin cabeza, capaces de agujerear la membrana de la bacteria, pero sin introducir su ADN”, explica Jaramillo.
Así, estas moléculas inducirían la muerte de la bacteria por la despolarización del citoplasma. “Al agujerear la membrana, se produce una diferencia de carga donde los iones se escapan, provocando la muerte de la bacteria”, relata el investigador del CSIC. “No hay resistencia bacteriana conocida contra este efecto”, sostiene. Su equipo pretende desarrollar estas moléculas combinando ingeniería genética con evolución, gracias a una ayuda del programa de investigación de la Fundación ‘La Caixa’ de cerca de medio millón de euros.
El equipo de investigación del I2SysBio pretende usar la evolución para crear moléculas antimicrobianas basadas en las proteínas que producen los fagos para insertar su ADN en las bacterias. Para ello, van a desarrollar una tecnología capaz de acelerar la evolución de fagos un millón de veces, permitiendo obtener fagos sin cabeza (cápside). Además, permitirá anticipar las mutaciones que podrían hacer resistentes las bacterias y adaptar así las moléculas antimicrobianas a esas mutaciones.
Así, los antibacterianos que desarrollarán gracias a este proyecto son meras agrupaciones de proteínas, no virus. No se pueden replicar, ni en la bacteria, ni en nuestro propio organismo, y serán inocuos para las bacterias beneficiosas, lo que resolverá uno de los efectos indeseados de los antibióticos actuales.
Según Jaramillo, esta estrategia mantiene las ventajas de la terapia con fagos que se aplica hoy día contra las RAM, pero permite obtener antimicrobiales que evitan las posibles resistencias de la bacteria al fago. Además, al tratarse de moléculas que, contrariamente a los fagos, no evolucionan y además no son organismos modificados genéticamente, su autorización sanitaria resultaría más sencilla. También se trataría de un método más rápido y barato, puesto que las moléculas se obtendrían por fermentación en biorreactores.
El proyecto, que tiene una duración de 3 años a partir de enero de 2023, quiere demostrar que esta tecnología es útil y viable para la producción de agentes antimicrobianos.








 Lilisbeth Perestelo:
Lilisbeth Perestelo:  César Hernández, director general de Cartera y Farmacia del Ministerio de Sanidad:
César Hernández, director general de Cartera y Farmacia del Ministerio de Sanidad:  Kilian Sánchez, secretario de Sanidad del PSOE y portavoz de la Comisión de Sanidad del Senado.:
Kilian Sánchez, secretario de Sanidad del PSOE y portavoz de la Comisión de Sanidad del Senado.:  Rocío Hernández, consejera de Salud de Andalucía:
Rocío Hernández, consejera de Salud de Andalucía:  Nicolás González Casares, eurodiputado de Socialistas & Demócratas (S&D - PSOE):
Nicolás González Casares, eurodiputado de Socialistas & Demócratas (S&D - PSOE):